Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista Portuguesa de Imunoalergologia
versão impressa ISSN 0871-9721
Rev Port Imunoalergologia vol.21 no.3 Lisboa set. 2013
Perfil de diversidade da comunidade estafilocócica da pele em doentes com dermatite atópica
Diversity profile from the skin staphylococcal community on atopic dermatitis patients
Cristina Lopes1,2, José Soares3, Freni Tavaria3, Rosa Silva4, Vera Oliveira4, José Morgado4, Luís Delgado1,5, Manuela Pintado2
1 Serviço & Laboratório de Imunologia, Faculdade de Medicina da Universidade do Porto
2 Unidade de Imunoalergologia, Hospital Pedro Hispano, Unidade Local de Saúde de Matosinhos
3 CBQF – Escola Superior de Biotecnologia, Universidade Católica Portuguesa, Porto
4 Centro Tecnológico e das Indústrias Têxtil e do Vestuário de Portugal, Vila Nova de Famalicão
5 CINTESIS (Center for Research in Health technologies and information systems), Universidade do Porto
RESUMO
Introdução: A dermatite atópica (DA) é uma doença cutânea crónica imunologicamente mediada em que a maioria dos doentes está colonizada por Staphylococcus aureus (S. aureus) capaz de produzir vários factores de virulência. O S. aureus pode ser cultivado em cerca de 90% das lesões cutâneas e pode colonizar a pele de aspecto morfologicamente normal. Os estafilococos coagulase negativos (SCN) geralmente não produzem toxinas com actividade superantigénica, mas o seu papel patogénico na DA não pode ser excluído. Neste estudo, pretende-se caracterizar a comunidade estafilocócica da pele de doentes com DA e indivíduos saudáveis, assim como identificar factores de virulência nas espécies identificadas. Métodos: Todos os isolados estafilocócicos foram submetidos a análise numérica de factores de virulência. As espécies isoladas da pele de doentes com DA e indivíduos saudáveis foram submetidas a técnicas de identificação molecular por Multiplex-PCR para identificação de bactérias pertencentes às espécies S. aureus, S. epidermidis, S. capitis, S. hominis e S. haemolyticus através de fragmentos específicos de ADN de 700, 124, 208, 806 e 271 bp, respectivamente. Identificação complementar de cada isolado, previamente identificado por Multiplex-PCR e de 22 isolados não identificados foram realizados por sequenciação do gene sodA. Resultados: Nos doentes com DA isolaram -se estirpes de S. aureus, com 71 (36,2%), S. epidermidis com 59 (30,1%) e S. hominis com 54 (27,6%) isolados. Foi analisada a pele de indivíduos -controlo saudáveis com prevalência para o S. warneri com 10 (23,8%) isolados e S. saprophyticus com 9 (21,4%) isolados juntamente com mais seis espécies identificadas, i.e., S. epidermidis, S. aureus, S. capitis, S. hominis, S. haemolyticus e S. lugdunensis. A maioria das espécies de estafilococos foi coagulase negativo (158/238 isolados) e desoxirribonuclease negativos (161/238). Verificou-se maior biodiversidade na pele de indivíduos saudáveis, com 8 espécies identificadas, do que na pele de doentes com DA, com 4 espécies identificadas. Conclusão: Existe uma maior diversidade de espécies estafilocócicas em indivíduos saudáveis comparativamente aos doentes com DA na presente amostra. A predominância de SA na pele de doentes com DA evidencia a sua maior adaptação. A caracterização detalhada e o perfil de virulência para cada doente poderão ser úteis numa terapêutica antimicrobiana individualizada. A relação simbiótica versus antagonista entre os estafilococos comensais e SA deverá ser melhor esclarecida.
Palavras-chave: Dermatite atópica, estafilococos, factores de virulência.
ABSTRACT
Introduction: Atopic dermatitis (AD) is a chronically inflammatory immunological mediated skin disease where the majo rity of patients are colonized with Staphylococcus aureus (S. aureus) capable of producing a quantity of virulence factors. S. aureus can be cultured from 90% of skin lesions and can colonize normal -appearing skin in patients with AD. Coagulase negative staphylococci (CNS) generally do not produce such toxins with superantigenic activity, but their role in AD pathogenicity should not be ruled out. Our aim was to study the staphylococcal community from skin of AD patients and normal healthy individuals without skin disruption, as well as some of the virulence factors present in these isolates. Methods: All staphylococcal isolates were subject to numerical analysis of some essential virulence factors. The majority of the staphylococcal species were coagulase (158/238 isolates) and deoxyribonuclease (161/238 isolates) negative. The wild strains isolated from the skin surface of healthy and AD individuals were submitted to multiplex PCR to identify the bacteria belonging to the S. aureus, S. epidermidis, S. capitis, S. hominis and S. haemolyticus species by yielding specific DNA fragments of 700, 124, 208, 806 and 271 bp, respectively. Complementary identification of each isolate previously identified by multiplex PCR and remaining 22 unidentified isolates was performed by sodA gene sequencing. Results: The staphylococcal microflora of the skin was dominated by S. aureus (71/196 isolates) followed by S. epidermidis (59/196 isolates) species in AD patients. In healthy individuals a high prevalence of S. warneri (9/42 isolates) followed by S. aureus (5/42 isolates) and S. haemolyticus (5/42 isolates) species was detected. High biodiversity in the skin of healthy individuals with eight staphylococcal species identified; whereas in the skin of AD patients only four species were distinguished Conclusion: Our study highlighted the higher diversity of the staphylococcal community occurring in the skin of healthy individuals versus AD patients. The predominance of S. aureus on the skin of individuals with AD suggests high properties of adaptation of this species to the disease condition. Future perspectives include the detailed characterization of the staphylococcal community and virulence profile for each patient as a mean of tailor made therapeutic approach; the symbiotic versus antagonistic relation between commensals and pathogenic species of Staphylococcus should be further investigated.
Keywords: Atopic dermatitis, Staphylococcus, virulence factors.
INTRODUÇÃO
A pele é um ecossistema complexo e dinâmico habitado por uma grande variedade de microrganismos.
A barreira exercida pela pele é essencial para a sobrevivência, impedindo a perda de humidade e lesão por agentes infecciosos ou tóxicos. A pele humana possui uma microfl ora residente que pode diferir consideravelmente de acordo com o local do corpo humano, sendo que nenhum outro órgão tem uma microfl ora com uma composição tão específi ca como a pele1. A população principal da microfl ora residente consiste em espécies de SCN, Propionibacterium acnes e espécies de Malassezia. A estabilidade deste grupo de microrganismos é baseada num equilíbrio constante com o sistema imunológico2,3.
A dermatite atópica (DA) é uma doença cutânea inflamatória crónica imunologicamente mediada. Clinicamente caracteriza-se por surtos e remissões de lesões intensamente pruriginosas e de localização variável5. Nas últimas décadas a sua prevalência duplicou nos países industrializados6,7, acarretando custos socioeconómicos elevados.
A sua patogénese é complexa, resultando da acção de diversos factores ambientais (alergénios, agentes infecciosos) em indivíduos geneticamente predispostos. Mutações no gene que codifica a filagrina, uma proteína do citoesqueleto da queratina, estão associadas à DA, particularmente em doentes que subsequentemente desenvolvem rinite e/ou asma alérgicas8. Ao contrário da pele de indivíduos saudáveis, a pele dos doentes com DA apresenta deficiências do sistema imunológico inato, quer celular (neutrófilos, células dendríticas plasmocitóides), quer de mediadores solúveis (quimiocinas envolvidas no recrutamento de linfócitos T, péptidos antimicrobianos, entre outros)4.
Estes doentes apresentam maior predisposição para colonização e infecção por S. aureus (em zonas com ou sem lesão aparente) que pode exacerbar o estado da doença9. Embora tradicionalmente o S. aureus tenha sido considerado um colonizador normal da pele em indivíduos saudáveis, estudos recentes10 parecem discordar desta afirmação. A patogenicidade do S. aureus é atribuível aos potenciais factores de virulência, como: proteínas de superfície que promovem a sua colonização, factores de superfície que inibem a fagocitose por células imunitárias, exotoxinas que danificam tecidos do hospedeiro (enterotoxinas estafilocócicas A-G, toxina da síndrome do choque tóxico, exfoliatinas) e resistência inerente ou adquirida a agentes antimicrobianos11.
A topografia da pele e respectivos microambientes são determinantes importantes da estrutura da comunidade microbiana em locais específicos. A DA envolve preferencialmente a região antecubital e poplítea – locais que abrigam grupos semelhantes de organismos mas com composições distintas no seio da comunidade microbiana residente10. Estes resultados sugerem que as comunidades microbianas podem contribuir para as predilecções de algumas patologias cutâneas em locais estereotipados. A caracterização da comunidade estafilocócica residente na pele de indivíduos com DA é fundamental para esclarecer a sua relevância na patogénese da doença. O objectivo deste trabalho foi caracterizar numa perspectiva molecular a biodiversidade da comunidade estafilocócica residente na pele e alguns factores de virulência associados em doentes com DA e sua comparação com indivíduos saudáveis.
MATERIAL E MÉTODOS
Amostra populacional
Foram incluídos 6 doentes (com idades entre 5 e 35 anos) com diagnóstico médico de DA moderada a grave com lesões de DA no momento da observação mas sem evidência clínica de infecção, sem tratamento com corticoides tópicos, inibidores da calcineurina, imunossupressores sistémicos ou antibióticos nas duas semanas prévias. Cinco dos doentes eram atópicos, sensibilizados a alergénios inalantes (ácaros e pólenes de gramíneas), três dos quais com diagnóstico prévio de asma. Um dos doentes apresentava concomitantemente alergia alimentar (anafilaxia a frutos secos). Foram incluídos 21 indivíduos saudáveis (com idades entre 18 e 24 anos) sem diagnóstico prévio de doenças do foro dermatológico, rinite e asma alérgica.
Colheita das amostras
Foram colhidas amostras da fossa poplítea e antecubital nos dois grupos, sendo que nos doentes com DA foram recolhidas amostras em locais de lesão de DA (13 amostras).
As amostras foram colhidas em dias independentes e transportadas imediatamente para o laboratório sob refrigeração. No dia de recolha das amostras não foi requerido aos indivíduos analisados nenhuma preparação prévia da pele. A superfície total de pele amostrada em cada zona foi de 25 cm2 utilizando a técnica da zaragatoa.
As amostras recolhidas foram inoculadas em Baird Parker Medium (Lab M, Lancashire, Reino Unido) a 37 °C durante 24 a 48 horas.
Estirpes de referência
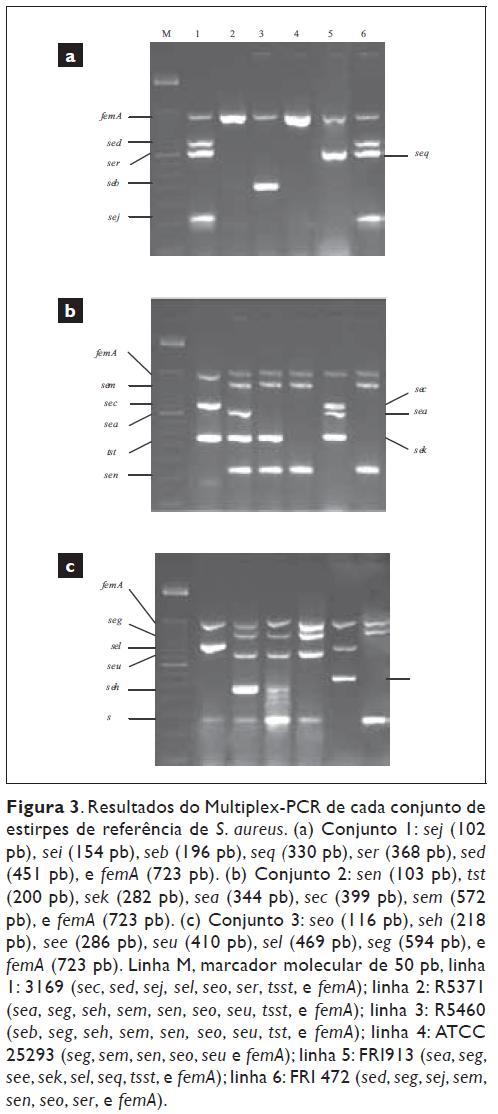
As estirpes de referência utilizadas no Multiplex-PCR foram: S. aureus ATCC 25923, S. epidermidis ATCC 14990, S. capitis subsp. capitis ATCC 27840, S. hominis subsp. Hominis ATCC 27844 e S. haemolyticus ATCC 29970. Os controlos positivos utilizados na detecção dos genes produtores de toxinas, a partir de estirpes referência de S. aureus foram: R5371/00 (SEA, SEG, SEH, SEI, SEM, SEN, SEO, SEU e TSST -1), R5460/00 (SEB, SEG, SEH, SEM, SEN, SEO, SEU e TSST -1), FRI 472 (SED, SEG, SEJ, SEM, SEN, SEO e SER), 3169 (SEC, SED, SEJ, SEL, SEO, SER e TSST-1) e FRI 913 (SEA, SEC, SEE, SEL, SEK, SEQ e TSST-1) fornecidas gentilmente pelo Prof. Løvseth (Secção de Alimentação e Microbiologia de Alimentos, Veterinária Nacional Institute, Ullevålsveien, Oslo, Noruega).
Identificação molecular
Multiplex -PCR: A colecção dos isolados totais foi submetida à tecnologia de Multiplex -PCR. O ADN celular foi isolado pelo método da guanidina-isotiocianato como descrito11.
O processo de amplificação foi efectuado num termociclador Techne TC -512 (Cambridge, Reino Unido) usando um volume total de reacção de 25 μL contendo 4 μL de ADN (50 ng/μL), 2,5 μL de uma mistura de 5 primers (0,5 μM cada primer), 12,5 μL da NZYTaq 2x Green Master Mix e 6 μL de água estéril. Os primers utilizados na reacção de Multiplex e os produtos de amplificação obtidos estão descritos no Quadro 1. As condições de amplificação foram: desnaturação inicial à temperatura de 94 ºC durante 2 minutos e 30 segundos, seguido de 35 ciclos de amplificação a 94ºC durante 1 minuto, 50ºC durante 1 minuto e 15 segundos e 72ºC durante 1 minuto, ao que se seguiu uma extensão final a 72ºC durante 5 minutos.
Os produtos PCR resultantes foram analisados por electroforese em gel de agarose a 2%.
Sequenciação do gene sod A: Os isolados não identificados por Multiplex foram identificados por sequenciação parcial do gene sodA, como descrito12.
Factores de virulência
Testes fenotípicos: O teste da coagulase foi realizado por inoculação de cada isolado em 0,5 mL de plasma de coelho (Biomérieux, Marcy -lEtoile, França) e incubação a 37°C com leitura de coagulação a 30 minutos e 24 horas. A enzima desoxirribonuclease (DNase) foi detectada em meio gelose DNase (Pronadisa, Madrid, Espanha) com incubação a 37°C durante 24 horas. De forma a determinar o tipo de hemólise, inoculou-se cada estirpe em meio gelose sangue de carneiro (Biomérieux) e as placas foram incubadas a 37°C durante 24 horas.
Detecção de enterotoxinas por Multiplex-PCR: os primers usados para as enterotoxinas e a toxina da síndrome do choque tóxico e as condições do Multiplex-PCR foram preparados como descrito em Hwang et al.5.
RESULTADOS
Identificação molecular
Após isolamento das estirpes das placas de Baird Parker Medium foram obtidos uma colecção total de 238 isolados, dos quais 196 e 42 foram provenientes de indivíduos com DA e indivíduos saudáveis, respectivamente.
A colecção dos isolados totais foi avaliada por tecnologia de Multiplex -PCR (Figura 1). A identificação molecular baseou-se na amplificação de sequências específicas para as espécies-alvo: S. aureus, S. epidermidis, S. capitis, S. hominis e S. haemolyticus, resultando em produtos PCR de 723 bp, 124 bp, 208 bp, 866 bp e 271 bp, respectivamente.
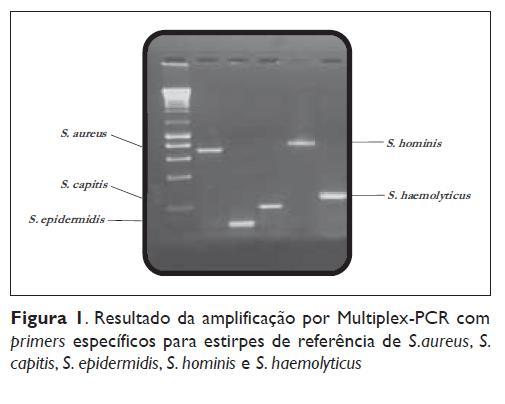
Todos os primers amplificaram com sucesso os genes-alvo na reacção de PCR sem produção de bandas inespecíficas.
A tecnologia de Multiplex implementada permitiu identificar 216 (90,7%) dos isolados totais. Os restantes isolados (n=22) foram identificados por sequenciação parcial do gene sodA.
O padrão total de identificação das estirpes isoladas está descrito na Figura 2. As estirpes prevalentes em doentes com DA foram o S. aureus, com 71 (36,2%) isolados, seguido por S. epidermidis com 59 (30,1%) e S. hominis com 54 (27,6%).
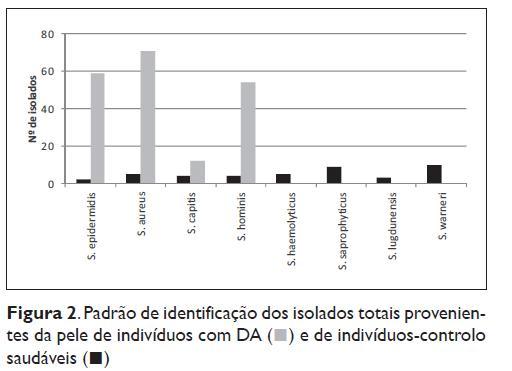
Em indivíduos saudáveis foi encontrado uma maior biodiversidade de espécies com um total de 8 espécies identificadas. S. epidermidis, S. aureus, S. capitis, S. hominis, S. haemolyticus, S. saprophyticus, S. lugdunensis e S. warneri (Figura 2). Dessas espécies, as mais prevalentes foram o S. warneri com 10 (23,8%) e o S. saprophyticus com 9 (21,4%) isolados.
Na DA nota-se uma redução da heterogeneidade no número de espécies identificadas (n=4) e paralelamente um aumento significativo na proporção de cada uma das espécies identificadas.
Factores de virulência
Detecção das enterotoxinas: Foi utilizado um ensaio de Multiplex-PCR (Figura 3) que permitiu a detecção dos 19 genes que codificam as enterotoxinas estafilocócicas (SEA-SEE, SEG -SER e SEU) e a toxina da síndrome do choque tóxico5.
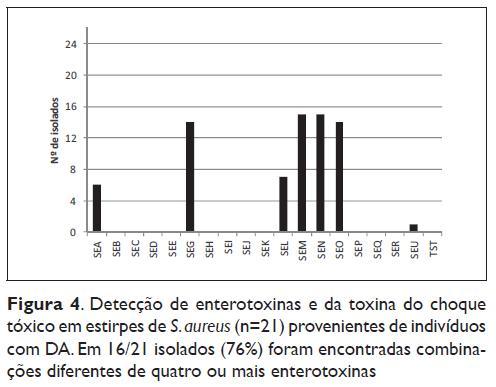
Os resultados da detecção dos genes das enterotoxinas e da toxina da síndrome do choque tóxico estão apresentados na Figura 4. Na detecção dos genes produtores de toxinas foram avaliados 21 dos isolados provenientes de doentes com DA e identificados com S. aureus. Foram encontradas 7 enterotoxinas diferentes nos 21 isolados. Em 76% (16/21) dos isolados foi detectada a presença de pelo menos um gene produtor de enterotoxina. As toxinas encontradas com maior frequência foram a SEG (14/21), SEM (15/21), SEN (15/21) e SEO (14/21), sempre encontradas juntas no mesmo isolado, com excepção de uma única estirpe. Neste estudo, foram ainda detectadas, mas com uma taxa menor de incidência, a SEA (6/21), SEL (7/21) e SEU (1/21). De acordo com o seu perfil genético, foram encontrados 7 genótipos com combinações diferentes de enterotoxinas (resultados não apresentados).
Testes fenotípicos: Incluíram a detecção da actividade de coagulase e DNase, assim como a avaliação da actividade hemolítica em placa de gelose sangue. A grande maioria dos isolados (68,1%) demonstrou ser coagulase e DNase negativo. No que diz respeito à actividade hemolítica, 67% dos isolados não apresentaram actividade hemolítica e 33% demonstraram β-hemólise. Não foi detectada α-hemólise nos isolados totais.
DISCUSSÃO
A DA é uma doença inflamatória cutânea classicamente associada à colonização e infecção por um grupo de microrganismos específicos. A caracterização detalhada do microbioma cutâneo nesta patologia é fundamental para uma abordagem terapêutica adequada.
No presente estudo, a metodologia molecular permitiu caracterizar de forma eficaz e abrangente o perfil da microflora estafilocócica residente na pele. A microflora identificada foi distinta nos dois grupos, notando-se uma heterogeneidade nas espécies identificadas entre os indivíduos com DA e indivíduos saudáveis.
Verificou-se uma redução da diversidade bacteriana nos indivíduos com DA relativamente aos indivíduos saudáveis com 4 e 8 espécies identificadas (Figura 2), respectivamente.
A DA tem sido associada a uma prevalência elevada de colonização da pele por estirpes de S. aureus, o que está intimamente relacionado com a gravidade da doença8.
O S. aureus foi a estirpe prevalente no contexto da DA com 71/196 isolados. Do grupo de 6 indivíduos analisados com DA, 4 (67%) estavam colonizados por S. aureus. Outros autores13,14 demonstraram que até cerca de 80% dos doentes com DA podem estar colonizados por S. aureus. No grupo de 21 indivíduos saudáveis, 5 (24%) estavam colonizados por estirpes de S. aureus, resultados que estão de acordo com o previamente descrito15.
Também ocorreram flutuações em outras espécies de bactérias presentes na pele. O S. epidermidis e o S. homi nis são espécies comuns na pele de indivíduos saudáveis e têm sido considerados microrganismos comensais, com a capacidade de inibir o S. aureus16. A proporção destas duas espécies também aumenta consideravelmente nos indivíduos com DA, sugerindo uma visão diferente sobre a relação da comunidade estafilocócica com a DA. Estas espécies de estafilococos podem compartilhar uma relação mutualística ou comensal para melhorar a resistência comum a peptídeos antimicrobianos. Alternativamente também foi sugerido como sendo um mecanismo compensatório de aumento destas estirpes de Staphylococcus coagulase negativos com o intuito de controlar o S. aureus8.
Existem várias linhas de evidência apoiando o conceito de que as toxinas com capacidade de superantigénios desempenham um papel na patogénese da DA. Estes superantigénios podem penetrar a barreira da pele e contribuir para a persistência e exacerbação da inflamação alérgica da pele17. Do ponto de vista clínico, mais do que 50% dos doentes com DA são colonizados por estirpes de S. aureus que secretam, pelo menos, um superantigénio14.
Neste estudo, em 16/21 (76%) estirpes de S. aureus isoladas de indivíduos com DA foram detectadas exotoxinas, com combinações diferentes de quatro ou mais enterotoxinas. Estudos prévios demonstraram que 30 a 60% das estirpes de S. aureus isoladas de indivíduos com DA eram toxigénicas13,14. Os genes predominantes foram o SEM, SEM, SEG e SEO. Estes genes são conhecidos como componentes normais do operão egc (SEG, SEI, SEM e SEN com SEO ou SEU), de modo que este elemento genético móbil foi frequentemente detectado5 e pode ser considerado uma possível fonte de genes com actividade superantigénica. Mempel et al.18
também identificou a presença do operão egc em estirpes de S. aureus isoladas de doentes com DA. De notar ainda que das enterotoxinas clássicas (SEA -SED), apenas a SEA foi identificada em 6/21 (28,5%) isolados. Alguns estudos apontam a SEB19 ou a SEC20 como as toxinas preferencialmente associadas com a DA, no entanto, neste estudo não foram detectados os genes produtores destas toxinas. Esta caracterização deveria também ser alargada às estirpes de SCN isoladas neste estudo, de modo a verificar a taxa de incidência de estirpes de SCN toxigénicas nos isolados totais e realçar melhor o papel de estipes de SCN no contexto da DA.CONCLUSÕES
Existe uma maior diversidade de espécies estafilocócicas em indivíduos saudáveis comparativamente aos doentes com DA na nossa população. As estirpes prevalentes em doentes com DA foram o S. aureus, seguido do S. epidermidis e S. hominis. A relação entre as diversas espécies da comunidade estafilocócica cutânea é complexa e ainda não está completamente esclarecida. Poderá ter sentido no futuro imunomodular a pele com espécies comensais não patogénicas?
A possibilidade de estabelecer para cada doente com DA o seu perfil estafilocócico tem importantes e potenciais implicações clínicas: dirigir a terapêutica antimicrobiana de forma específica, tailor -made à comunidade microbiana daquele doente e não de forma empírica.
Em estudos futuros a caracterização do perfil de virulência de espécies SCN provenientes de doentes com DA poderá esclarecer o seu papel na manutenção da inflamação cutânea.
REFERÊNCIAS
1. Rosenthal M, Goldberg D, Aiello A, Larson E. Skin microbiota: microbial community structure and its potential association with health and disease. Foxman BInfect Genet Evol 2011;11:839-48. [ Links ]
2. Fredricks DN. Microbial ecology of human skin in health and disease. J Investig Dermatol Symp Proc 2001;6:167-9. [ Links ]
3. Katsuyama M, Ichikawa H, Ogawa S, Ikezawa Z. A novel method to control the balance of skin microflora. Part 1. Attack on biofilm of Staphylococcus aureus without antibiotics. J Dermatol Sci 2005;38:197-205. [ Links ]
4. Lopes C, Duarte AF, Correia O, Delgado L. Atopic dermatitis, innate immunity, and infection. Dermatol Online J 2011;17:4. [ Links ]
5. Hwang SY, Kim SH, Jang EJ, Kwon NH, Park YK, Koo HC, et al. Novel multiplex PCR for the detection of the Staphylococcus aureus superantigen and its application to raw meat isolates in Korea. Int J Food Microbiol 2007;117:99-105. [ Links ]
6. Asher MI, Montefort S, Björkstén B, Lai CK, Strachan DP, Weiland SK, et al. Worldwide time trends in the prevalence of symptoms of asthma, allergic rhinoconjunctivitis, and eczema in childhood: ISAAC Phases One and Three repeat multicountry cross -sectional surveys. Lancet 2006;368:733-43. [ Links ]
7. Shaw TE, Currie GP, Koudelka CW, Simpson EL. Eczema prevalence in the United States: data from the 2003 National Survey of Childrens Health. J Invest Dermatol 2011;131:67-73. [ Links ]
8. Kong HH, Oh J, Deming C, Conlan S, Grice EA, Beatson MA, et al. Temporal shifts in the skin microbiome associated with disease flares and treatment in children with atopic dermatitis. Genome Res 2012;22:850-9. [ Links ]
9. Boguniewicz M, Leung DY. Recent insights into atopic dermatitis and implications for management of infectious complications. J Allergy Clin Immunol 2010;125:4-13. [ Links ]
10. Grice EA, Segre JA. The skin microbiome. Nat Rev Microbiol 2011;9:244-53. [ Links ]
11. Cuny C, Witte W. Typing of Staphylococcus aureus by PCR for DNA sequences flanked by transposon Tn916 target region and ribo somal binding site. J Clin Microbiol 1996;34:1502-5. [ Links ]
12. Poyart C, Quesne G, Boumaila C, Trieu -Cout P. Rapid and accurate species – level identification of coagulase-negative staphylococci by using the sodA gene as a target. J Clin Microbiol 2001;39:4296-301. [ Links ]
13. Bunikowski R, Mielke ME, Skarabis H, Worm M, Anagnostopoulos I, Kolde G, et al. Evidence for a disease-promoting effect of Staphylococcus aureus -derived exotoxins in atopic dermatitis. J Allergy Clin Immunol 2000;105:814-9. [ Links ]
14. Nada RA, Shaheen HI, Touni I, Fahmy D, Armstrong AW, Weiner M, et al. Design and validation of a multiplex polymerase chain reaction for the identification of enterotoxigenic Escherichia coli and associated colonization factor antigens. Diagn Microbiol Infect Dis 2010;67:134-42. [ Links ]
15. Higaki S, Kitagawa T, Morohashi M, Yamagishi T. Distribution and antimicrobial susceptibility of coagulase -negative staphylococci from skin lesions. J Int Med Res 1999;27:191-5. [ Links ]
16. Iwase T, Seki K, Shinji H, Mizunoe Y, Masuda S. Development of a real-time PCR assay for the detection and identification of Staphylococcus capitis, Staphylococcus haemolyticus and Staphylococcus warneri. J Med Microbiol 2007;56:1346 -9. [ Links ]
17. Schlievert PM, Case LC, Strandberg KL, Abrams BB, Leung DY. Superantigen profile of Staphylococcus aureus isolates from patients with steroid-resistant atopic dermatitis. Clin Infect Dis 2008; 46:1562-7. [ Links ]
18. Mempel M, Lina G, Hojka M, Schnopp C, Seidl HP, Schäfer T, et al. High prevalence of superantigens associated with the egc locus in Staphylococcus aureus isolates from patients with atopic eczema. Eur J Clin Microbiol Infect Dis 2003;22:306-9. [ Links ]
19. Bonness S, Szekat C, Novak N, Bierbaum G. Pulsed -field gel electrophoresis of Staphylococcus aureus isolates from atopic patients revealing presence of similar strains in isolates from children and their parents. J Clin Microbiol 2008;46:456-61. [ Links ]
20. Tomi NS, Kränke B, Aberer E. Staphylococcal toxins in patients with psoriasis, atopic dermatitis, and erythroderma, and in healthy control subjects. J Am Acad Dermatol 2005;53:67-72. [ Links ]
Cristina Lopes
Serviço e Laboratório de Imunologia
Faculdade de Medicina da Universidade do Porto
Alameda Prof. Hernâni Monteiro
4200-450 Porto
E-mail: clabreu@med.up.pt
Financiamento: Estudo financiado pelo Projecto 2nd Dermis – IDT Individual, promovido pela empresa Crispim Abreu, cofinanciado no âmbito do Quadro de Referência Estratégico Nacional (QREN) segundo a Portaria 1462/2007 de 15 de Novembro.
Declaração de conflitos de interesse: Nenhum.
Nota: Prémio SPAIC – Bial-Aristegui 2012 (2.º Prémio)
Data de recepção / Received in: 31/07/2012
Data de aceitação / Accepted for publication in: 05/10/2012